
近日,课题组在抗菌肽从头设计领域取得新进展,于2023年2月1日在国际知名期刊《Briefings in Bioinformatics》在线发表了基于深度学习和分子动力学模拟实现抗菌肽的从头设计研究成果,这是课题组在利用深度学习实现多肽从头设计的首次尝试,为课题组开辟了新的研究方向。
近年来,抗生素的滥用导致大量多药耐药菌出现,对全球公共卫生安全构成了严重的威胁。抗菌肽由于具有较低毒性、广谱抗菌活性以及不易引起耐药性而备受关注,有希望取代传统抗生素治疗细菌感染。传统多肽药物设计往往依赖于个人的专业经验,整个过程耗时耗力。随着计算机技术的进步,人工智能和机器学习在药物发现中发挥着关键作用。目前,深度学习已应用于抗菌肽的从头设计,并能够高效地解决抗菌肽的分类问题。
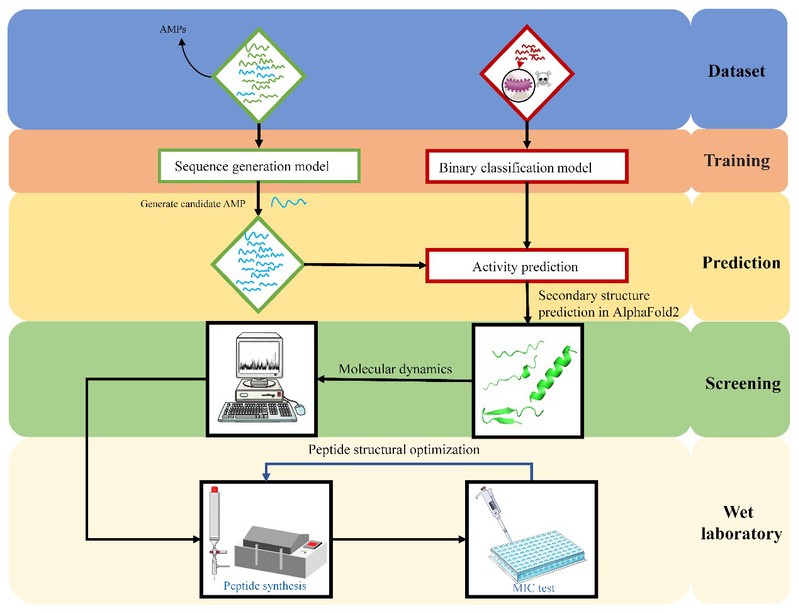
在这项研究中,研究人员结合了几种自然语言处理模型来设计和识别抗菌肽,即序列生成对抗网络 (SeqGAN)、BERT和多层感知器(MLP)。随后,研究人员将二级结构的存在及其稳定性作为进一步筛选候选抗菌肽的重要依据。最终,经过AlphaFold2和分子动力学结合,成功筛选出6条候选抗菌肽。其中,A-222对多种革兰氏阴性菌和革兰氏阳性菌表现出高效力。经过NMR结构解析、圆二色谱和AlphaFold2结构预测证明了A-222的α螺旋的存在。随后,研究人员设计并合成了A-222的新类似物,并通过丙氨酸扫描以及增加带正电残基的数目来评估对抗菌活性的影响。随后发现A-222的类似物针对嗜麦芽糖杆菌WH 006和铜绿假单胞菌PAO1的活性增加了4-8倍,并且几种类似物的MIC达到了8μg/mL,这与天然的抗菌肽活性相当。这项工作证实了深度学习和分子动力学模拟在加速抗菌肽发现方面的潜力。
课题组硕士研究生曹秋实,博士研究生葛成为文章共同第一作者,于日磊教授、江苏理工学院生物信息与医药工程研究所所长常珊教授以及中国海洋大学海洋生命学院王岩教授为文章共同通讯作者。该研究由国家自然科学基金等项目支持。
文章链接:https://doi.org/10.1093/bib/bbad058